Xylella fastidiosa : Etat des connaissances et axes de recherche - Philippe Reynaud (Anses Montpellier) avec l'aide de J.Y. Rasplus et al. (INRA) ...
←
→
Transcription du contenu de la page
Si votre navigateur ne rend pas la page correctement, lisez s'il vous plaît le contenu de la page ci-dessous
Xylella fastidiosa :
Etat des connaissances et axes
de recherche
Philippe Reynaud (Anses Montpellier) avec l’aide de J.Y.
Rasplus et al. (INRA) et F. Poliakoff et al. (Anses Angers)Anses - Laboratoire de la santé des végétaux
Laboratoires
Unité
de référence Unité
Nématologie Mycologie
Rennes Nancy
Siège
et
Unité Bactériologie,
virologie, OGM
Angers
Unité
Quarantaine
Clermont-
Ferrand
Unité Entomologie et
plantes invasives
Montpellier
Unité Ravageurs et
agents pathogènes
tropicaux
La RéunionXylella fastidiosa
• Bactérie du xylème
Cortex
Epiderme
Moelle
Cambium
Xylème Phloème
Xylème Phloème
CambiumModélisation du mouvement de X. fastidiosa dans le xylème
Bactérie
Signal chimique
Karyn L. Newman et al. PNAS 2004;101:1737-1742Découverte • “California Vine Disease”: Première détection à Anaheim (Ca) en 1884 (Pierce 1892) Newton B. Pierce
Découverte • 1949 : virus? (Hewitt et al.) • Premier isolement sur milieu en 1978 (Davis et al.) • Nommée en 1987 (Wells et al.) • 1987 : Chlorose panachée des Citrus au Brésil • Fin des années 1990 : maladie de Pierce, California
Xylella fastidiosa : distribution
Ontario
1880
Canada
USA
Mexico Italie
Costa Rica
Venezuela
Equateur
Brésil
Brésil
Paraguay 1987
ArgentineXylella fastidiosa : distribution
interceptions en 2012, 2014 et 2015
sur plants de caféier
Ontario
1880
Canada 2014
USA
2013 2013
Italie
Mexico Italie
Iran
Iran Taiwan
Costa Rica
Venezuela Foyers
Equateur
Brésil
Brésil
Paraguay 1987
ArgentineTaxonomie et spectre d’hôte
5 sous-espèces généralistes (env 300 plantes/60
familles au total)
Lignées génétiques avec spectres d’hôte plus
réduits (à confirmer)
Xf fastidiosa (PD, ALS) : vigne, amandier, 42
familles
Xf sandyi (OLS) : laurier rose, caféiers
Xf morus : muriers (fastidiosa x multiplex)
Xf multiplex (PDD, PLS) : Prunus, Quercus, oliviers,
Polygala etc
Xf pauca (CVC) : agrumes, oliviers, caféiers
PD = Pierce’s di sease
ALS = Almond leaf scorch di sease
PDD = Phony peach di sease
PLS = Plum leaf scorch
OLS = Oleander leaf scorch
CVC = Citrus variegated chlorosisX. fastidiosa : distribution des sous espèces
subsp. fastidiosa
subsp. multiplex
subsp. pauca
subsp. sandyi
Ontario
Canada
USA
Italie
Mexico
Italie
Iran
Iran
Costa Rica
Venezuela
Equateur
Brésil
Paraguay
Brésil
ArgentineIdentification des souches X. fastidiosa en France
2 profils différents de Xylella fastidiosa,
présents à la fois en Corse ST7
et en région PACA:
X. fastidiosa subsp. multiplex
profil A (ST7)
profil B (ST6)
Distinctes de la
souche CoDiRO
X. f. subsp. pauca ST6
présente en Italie
MLSA/MLST
(Scally et al., 2005 / Yuan et al., 2010)
11Les insectes vecteurs
Les espèces vectrices potentielles Aphrophoridae
4 familles d’Hémiptères (51 spp F, 119 UE)
Cicadidae Aphrophoridae (15 sp F, 29 sp UE)
Cercopidae (7 sp en F et UE) Aphrophora alni
Cicadellidae (9 sp en F et UE)
Cicadidae et Tibicinidae (20 sp en F et 74 en
UE)
Piqueurs-suçeurs de xylème
Mal connus
Philaenus spumarius
Globalement polyphages
Cercopidae
Pour la plupart communs
Lyristes plebejus Populations importantes
Déplacement relativement faible
En général une génération par an
Passe l’hiver sous forme d’œufs ou de
larves (cigales) Cercopis larve
Cicadella viridis Cercopis intermedia
Graphocephala fennahi
USA => UE
Cicada orni CicadellidaeCycle biologique type d’un Cicadellinae
Adulte
Œufs
Larves vectrices
Adultes vecteursLes vecteurs nord-américains
Piqueurs-suçeurs de xylème
Appartiennent à des genres et des groupes différents Graphocephala sp
Bien connus, mais connaissance difficilement transposable
Plusieurs générations par an en région chaude
Certaines espèces passent l’hiver à l’état adulte
Un vecteur introduit récemment en CA a eu un impact fort
sur la propagation de la maladie (Homalodisca vitripennis)
Draeculacephala sp
Philaenus spumarius UE => USA
Neophilaenus lineatus UE => USA
Homalodisca liturata
Cicadellidae
Draeculacephala minerva Graphocephala atropunctataXylème
Cibarium / précibariumX. fastidiosa ne passe pas d’un stade à l’autre
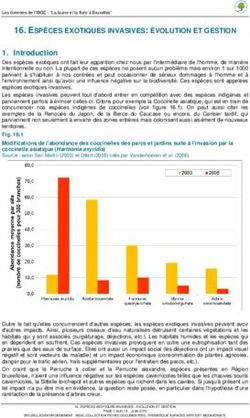
Plantes hôtes identifiées en Corse • Acer pseudoplatanus • Lavandula stoechas • Artemisia arborescens • Myrtus communis • Asparagus acutifolius • Hebe sp. • Cistus monspeliensis • Polygala myrtifolia • Cistus salviifolius • Prunus cerasifera • Coronilla valentina • Quercus suber • Cytisus racemosus • Rosa x floribunda • Genista ephedroides • Rosmarinus officinalis • Pelargonium graveolens • Spartium junceum • Lavandula angustifolia • Lavandula dentata En vert: nouvelles plantes hôtes (genre et/ou espèce), non listées précédemment par l’EFSA (Scientific opinion 2015). Scientific report EFSA 2016 : 359 espèces hôtes / 75 familles botaniques
Méthode de référence MA039 version 1
Détection de X. fastidiosa sur plantes hôtes par PCR en temps réel :
Méthode évaluée et validée
Méthode disponible sur : www.anses.fr
Extraction d’ADN automatisée avec le kit QuickPick™ Plant DNA (Bio-
Nobile) et l’automate KingFisher™ mL
PCR en temps réel Harper et al., 2010, Erratum 2013Méthode de référence MA039 version 1
Xylella fastidiosa: bactérie du xylème
Analyse réalisée sur tissus fortement vascularisés de
plantes en végétation: pétioles et/ou nervures centrales
0,5 à 1 g par
échantillon:
1 échantillon soit 5 à 100 pétioles
2 prélèvements pour
extraction d’ADN
4 amplifications géniques• Etat des connaissances • Axes de Recherche
La problématique Xylella est complexe
Cultures
Prunus
Olive
?
?
Co
Vecteurs Ps ?
?
Cs
? Xfp
Xfm
Réservoirs Maladie
AsymptomatiqueAxes de Recherche
Identifier les vecteurs, diagnostiquer la
bactérie et identifier la souche concernée
Travaux
d’identification des Test de
souches de Xylella pathogénicité
Travaux pour la
détection de Xylella
sur les vecteurs Prévision et gestion
du risque par la
modélisation
Surveillance des vecteurs potentiels
Evaluation des types de piégeage
Collection de référence
Création de clés d’identifications
26Anses – INRA : Travaux d’identification des souches de Xylella
Identification des sous-espèces par
MLSA
Phylogénie par MLST
Séquençage partiel des gènes
CysG, gltT, holC, leuA, malF, nuoL, petC
Centre International
de Ressources
Microbiennes
Xylella fastidiosa est très difficile à isoler de plantes contaminées. L’unité BVO (Angers) a néanmoins
réussit à isoler sur milieu artificiel :
17 souches sur échantillons de Corse
3 souches sur échantillons de PACA
7 souches sur caféiers interceptés
Afin d’identifier rapidement les sous-espèces en présence sans réaliser d’isolement, l’Unité BVO a
optimisé les PCR Hernandez-Martinez et al. (2007) et Pooler et Hartung (1995) permettant de discriminer
les quatre principales sous-espèces (fastidiosa, multiplex, pauca, sandyi), confirmées par MLSA
(séquençage de 7 gènes de ménage) (collaboration avec équipe INRA (Emersys).
4INRA/Anses : Test de pathogénicité : Projet régional SapAlien
Test de pathogénicité en cours (programme sur deux ans 2015- 2017)
Plantes hôtes potentielles
• Olea europaea Souches de X. fastidiosa
• Vitis vinifera • X. f. subsp. multiplex ST6 (Corsica)
• Nerium oleander • X. f. subsp. multiplex ST7 (Corsica)
• Coffea arabica • X. f. subsp. pauca (CoDiRO)
• Malus domestica • X. f. subsp. pauca (from intercepted
• Polygala myrtifolia Coffee tree)
• X. f. subsp. fastidiosa/sandyi (from
intercepted Coffee tree)
• X. f. subsp. fastidiosa (USA)
• X. f. subsp. multiplex (USA)
• Medicago sativa
• Nocotania tabacum
• Catharanthus roseus
9Anses : Travaux pour la détection de Xylella sur les vecteurs
Optimisation, évaluation et validation de méthodes moléculaires pour la
détection sur insectes (ex: Philaenus spumarius)
Janvier - mars 2016: Démarrage de l’optimisation de la méthode
Comparaison de méthodes d’extraction d’ADN
Utilisation de la PCR en temps réel (Harper et al., 2010)
Premiers résultats: bonne sensibilité avec le kit d’extraction ADN QuickPick
Plant DNA
Besoin d’insectes contaminés (d’Italie) pour valider la méthode (collecte
possible en avril)
Evaluation de méthode sur autres insectes vecteurs potentiels (cicadelles)
Second semestre 2016: test de la méthode NGS proposée par l’UMR CBGP (JY.
Rasplus)
Comparaison de méthodes d’analyses sur échantillons artificiellement
contaminés: NGS et PCR en temps réel (Harper et al., 2010), optimisation.
Test sur échantillons naturellement contaminés
Préparation d’un projet CASDAR pour application de ces méthodes à l’étude
comportementale des vecteurs potentiels sur cultures d’intérêt (novembre
2016)INRA : Identifier les vecteurs de Xylella, diagnostiquer la présence de la
bactérie et identifier la souche concernée par méthode NGS
Utilisation d’approches haut-débit
1 run de séquençage => 20 M de séquences
• 1 marqueur pour l’identification du vecteur
• 1 marqueur pour détecter toutes les
bactéries portées par le vecteur
• 1 ou 2 marqueurs pour identifier la plante
d’alimentation
• Les 7-9 marqueurs diagnostiques de Xylella
Jusqu’à 1500 individus sur une douzaine de marqueurs.
On peut savoir qui est qui, qui porte quoi, qui a mangé quoi
(applicable sur les plantes)
Recherche du/des vecteurs potentiels de Xylella fastidiosa :
Faible prévalence, vecteurs inconnus => nécessité du volumique
• Identifier qui porte quoi dans une communauté de vecteurs ?
• Assurer la biosurveillance du vecteur et du vecté à haut-débit
• Mesurer la prévalence du vecté dans des populations de vecteurs
• Identifier des liens entre microbiome et phénotype ou vectéINRA : Prévision et gestion du risque par la modélisation
Comprendre la diffusion Xylella (dispersion, climat, paysage).
Cartographie des zones à risque potentiel.
Informations Informations Modèle
écologiques climatiques statistique
Cicadelle
Homalodisca
vitripennis défavorable
sud USA, puis
Californie Estimation
vecteur de Xylella
fastidiosa
Maladie de Pierce
favorableAnses : projet H2020 POnTE (Pest Organisms Threatening Europe)
Collection de souches, Identification des
pathogénicité sur Citrus – vecteurs effectifs et
Caféier – Pervenche, Luzerne et proposition de
plantes ornementales, étude de la méthodes
colonisation des plantes hôtes d’échantillonnage
Ateliers pour
améliorer la capacité
des partenaires à la
détection précoce
d’émergence de
Xylella fastidiosa
Génétique de Xylella
Validation d’anticorps
pour l’IF, technique
d’extraction d’ADN
par automate sur
plantes et insectes,
validation de PCR
pour identification
des souches (RT
PCR, HRM)
Projet H2020 : Novembre 2015-Novembre 2019Anses : projet Euphresco VECTACROP
WP 2 : Surveillance des
vecteurs potentiels dans et
autour des cultures cibles
Evaluation des types de piégeage
Collection de référence des
vecteurs potentiels
Création de clés d’identifications
VECTACROP : Tracking vectors of
bacteria and phytoplasmas threatening
Europe’s major crops
WP 3 : Recherche de Xylella
dans les insectes (avec l’aide de
l’Anses Angers)
Projet : Mars 2016 - Mars 2018Merci
Vous pouvez aussi lire